DISEÑO DE MEDICAMENTOS
Ciencia de datos y reutilización de medicamentos

La fase de investigación y desarrollo de nuevos fármacos es un proceso largo y costoso, con un elevado porcentaje de fracasos. De hecho, se calcula que, por término medio, de cada 10.000 moléculas cribadas, sólo una dará lugar a una autorización de comercialización (AC), sabiendo que su desarrollo habrá durado entre 10 y 15 años y habrá costado, por ejemplo, en oncología, de 1.500 a 2.600 millones de dólares.
Una de las alternativas menos costosas en tiempo y dinero es el «drug repurposing». Esta metodología consiste en encontrar una nueva indicación terapéutica para sustancias farmacológicas cuyo desarrollo se ha interrumpido o que no han sido aprobadas en su indicación inicial.
Un ejemplo muy concreto de la aplicación de este método es la pandemia mundial vinculada al COVID19 en los últimos años. En efecto, mientras se esperaba la aprobación para la comercialización de vacunas de ARN mensajero u otras tecnologías biomédicas, la reutilización de medicamentos tuvo su lugar para encontrar tratamientos ya existentes que limitaran los efectos del virus SARSCOV2 en las personas infectadas.
Ante la gran cantidad de datos disponibles en fuentes abiertas o privadas, la principal dificultad estriba en cribar un gran número de sustancias farmacológicas en estas bases de datos y explicar de forma racional los resultados obtenidos. La ciencia de datos es, por tanto, la herramienta adecuada para resolver este tipo de problemas.
Objetivo del proyecto
En este proyecto, nuestro objetivo era reposicionar moléculas específicas en la lucha contra el SARS COV2. Para ello, tuvimos que identificar dianas farmacológicas basadas en los distintos mecanismos de acción del ciclo vírico del coronavirus y, a continuación, seleccionar moléculas con autorización de comercialización válida y un efecto conocido sobre las dianas previamente identificadas.
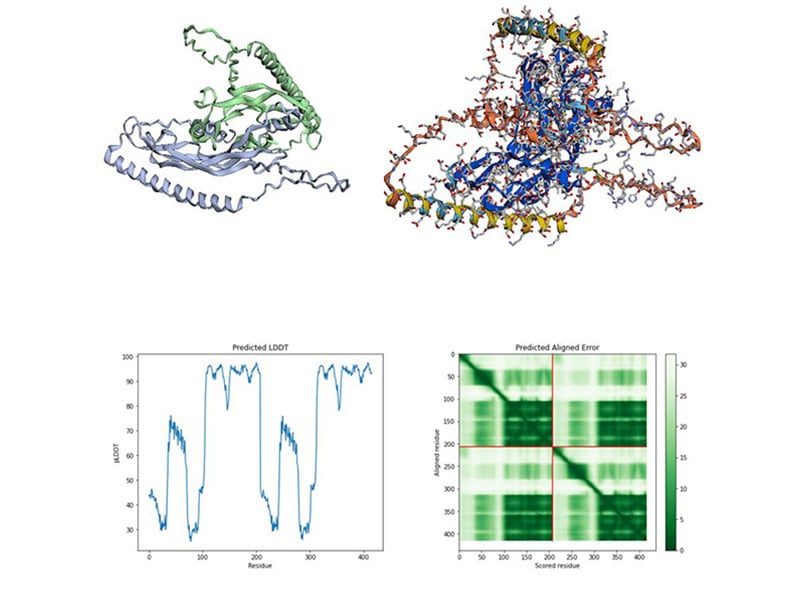
Un proyecto de colaboración
Contexto farmacológico
Para alcanzar nuestro objetivo, hemos trabajado en particular sobre los siguientes elementos
• Identificación de socios de interacción para una diana terapéutica implicada en una vía fisiológica determinada.
• Identificación del sitio o sitios activos que pueden inducir una respuesta farmacológica.
• Selección de moléculas con homología estructural.
• Clasificación de las moléculas identificadas en función de variables termodinámicas y de reacción.
• Selección de candidatos con un M.M.A. actualmente validado por las organizaciones sanitarias.
Resultados y conclusión
Identificamos 3 moléculas principales:
• VX-702
• Dilmapimod
• Binimetinib
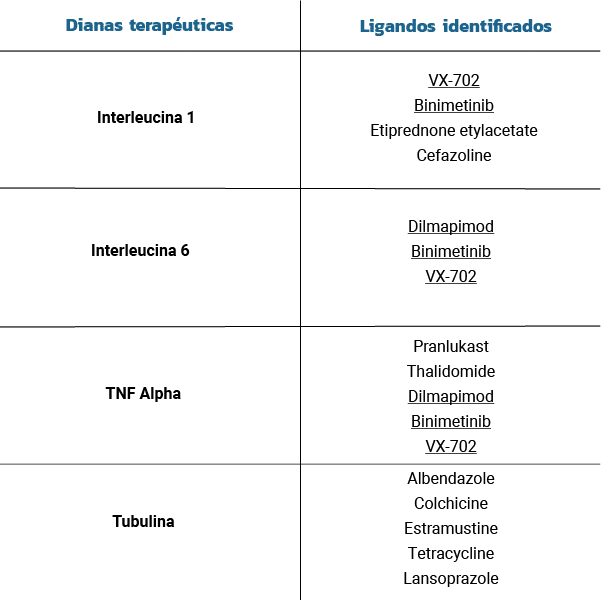
- Confirmamos nuestro modelo sabiendo que la mayoría de las moléculas identificadas habían sido o estaban siendo estudiadas en ensayos clínicos en el momento del proyecto.
- Intentamos explicar racionalmente los resultados obtenidos gracias a la literatura médica y farmacológica y planteamos así nuevas hipótesis de investigación sobre ciertas moléculas aún no estudiadas en la lucha contra el COVID19.